30 años de R+D CSIC
El coste de secuenciar genomas cuesta un millón de veces menos que hace algo más de dos décadas. El dato impresiona. Si en el año 1998, la secuenciación de un único trozo de genoma de la planta Arabidopsis fue un hito, ahora las cosas han cambiado radicalmente. Hablamos con algunos de los primeros investigadores en esta área.
 Pere Puigdomènech, en uno de los invernaderos del CRAG
Pere Puigdomènech, en uno de los invernaderos del CRAG
“Ahora se publican múltiples genomas a la vez”, apunta Pere Puigdomènech, profesor de investigación del CSIC. “El coste de la secuenciación se ha dividido por un millón, gracias a las técnicas de secuenciación masiva y al tratamiento de datos por computadora. Es un enorme avance tecnológico y nuestro conocimiento ahora es infinitamente mayor”.
Tal como explica este científico, ahora se dispone de herramientas para saber qué bases se quiere modificar, porqué y en qué parte del genoma, y éstas empiezan a ser usadas de forma general en las empresas del sector.
Puigdomènech es investigador emérito del CSIC en el Centro de Investigación en Agrigenómica (CRAG), un consorcio del CSIC, el IRTA, y las universidades UAB y UB. El origen del centro se remonta al año 2003, aunque el edificio no fue inaugurado hasta el año 2011. Sus instalaciones de 9000 metros cuadrados acogen invernaderos, cámaras de cultivo, laboratorios y oficinas para un equipo de investigadores y equipo técnico de más de dos centenares de personas, que investigan con el objetivo de comprender los carácteres demandados y esenciales de las plantas en relación con la alimentación.
Los inicios
El origen del CRAG, el primer centro de su tipo en España, se forjó en los laboratorios del CSIC y del IRTA. En el caso del CSIC, en el área de biología del Centro de Investigación y Desarrollo (CID). Entonces era un equipo de apenas una decena de personas, en el que se integró el mismo Puigdomènech, quien había vuelto de Alemania en 1981.
En 1985, publicaron la secuenciación de una proteína del maíz, la glutelina-2; el trabajo fue pionero en España y la primera secuencia de ADN publicada desde Cataluña. Actualmente, se publican varios genomas a la vez.
“Empezamos entonces a trabajar en ADN recombinante en plantas. Se unió a nosotros un grupo del CSIC que estaba localizado en la Universidad de Barcelona y que trabajaba en la mejora del maíz”. Entre 1982 y 1983 abordaron la secuenciación de una proteína del maíz, la glutelina-2, trabajo que se publicó en 1985. El trabajo fue pionero en España y la primera secuencia de ADN publicada desde Catalunya. Desde aquellas primeras publicaciones, los resultados se han acelerado de forma progresiva.
Ejemplos de una progresión acelerada
En la década de los 90, investigadores del CSIC que estaban entonces en el Centro de Investigación y Desarrollo (CID), participaron junto a un consorcio europeo en la secuenciación de un primer trozo del genoma de Arabidopsis, publicado en 1998. Arabidopsis es, desde hace años, el principal modelo de laboratorio por razones prácticas, como la sencillez y la facilidad de manipulación. De ahí el interés en obtener su genoma.
El genoma completo de Arabidopsis se publicó en el año 2000 (2), casi al mismo tiempo que el genoma humano. Participaron en el trabajo los investigadores del CSIC, junto a equipos de Europa, Estados Unidos y Japón. El proyecto, lanzado desde Bruselas en 1991, supuso una inversión de 100 millones de euros, aportados por la entonces Comunidad Europea, la National Science Foundation (EEUU) y el Instituto Kazusa (Japón). Los resultados, publicados en Nature, revelaron que esta planta herbácea tiene un genoma parcial de 119 millones de pares de bases (Mb) que contienen unos 26000 genes.
 Salomé Prat, investigadora del CSIC en el CRAG
Salomé Prat, investigadora del CSIC en el CRAG
Otro hito del mismo grupo, entonces ya en el Instituto de Biología Molecular integrado en el CID, fue descubrir en 2001 la proteína armadillo de patata, la cual se moviliza al núcleo en presencia de giberelinas y que más tarde dio pie a identificar cómo funcionan los represores DELLA, proteínas con una actividad clave en la señalización de estas hormonas. El trabajo fue liderado por Salomé Prat, profesora de investigación del CSIC, y apareció en la prestigiosa revista Cell (3). "Este segundo descubrimiento fue clave para entender por qué las plantas muestran un aspecto más alargado en ausencia de luz o también el motivo de la correlación existente entre una mayor tolerancia a estrés y la inhibición del crecimiento de la planta", explica la investigadora.
En 2012 se obtuvo el genoma completo del melón (4), de unos 450 millones de pares de bases y 27.427 genes. Lo hizo un consorcio español en el proyecto Melonics, dirigido por Pere Puigdomènech (CSIC), Jordi Garcia Mas (IRTA), y la colaboración de 14 grupos de investigación de diversos territorios de España y cinco empresas. La financiación del proyecto, impulsado por la Fundación Genoma España, ascendía a algo más de cuatro millones de euros: 25 veces menos que el coste de obtener el genoma de Arabidopsis doce años antes.
Si en septiembre de 2001, el coste de secuenciar un genoma se estimaba en algo más de 95 millones de dólares, en mayo de 2022, ese coste se había reducido a 525 dólares. Los costes de secuenciación de ADN en plantas son muy similares.
Reducción en el coste de secuenciar un genoma
Esa aparición constante y progresiva de resultados, genoma a genoma, contrasta con la situación actual en la que la tecnología ha avanzado tanto y tan rápidamente que ahora se puede obtener en poco tiempo y simultáneamente los genomas de distintas variedades. Un ejemplo: en 2021 se publicó en Science los genomas de 26 líneas de maíz, obtenidos simultáneamente en sólo dos años gracias a la tercera generación de secuenciación, conocida como “long-read sequencing”. No obstante, los autores reconocían el mérito a trabajos anteriores: todo ello no habría sido posible, decían, sin el primer genoma del B73, obtenido en 2009 (apareció en Science y fue un proyecto de cuatro años con una inversión de 31 millones de dólares, algo más de 22 millones de euros).
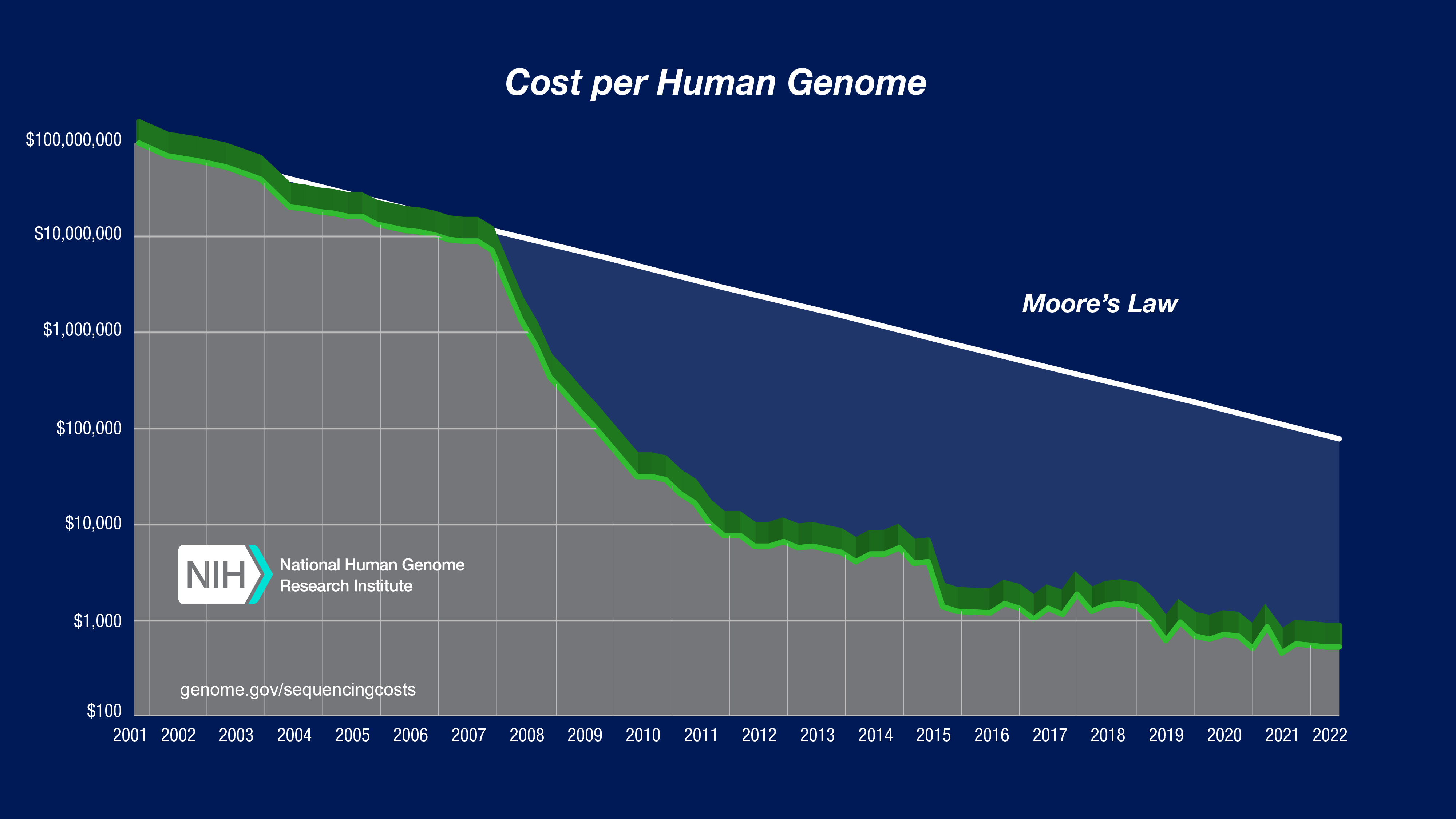 El Instituto Nacional de Investigación del Genoma Humano (NHGRI) de EE.UU. lleva mucho tiempo controlando los costes de la secuenciación de ADN realizada en centros de secuenciación financiados. Los últimos datos publicados en 2022 mostraban una drástica reducción de los costes. En septiembre de 2001, el coste de secuenciar un genoma se estimaba en algo más de 95 millones de dólares, coste que, en mayo de 2022, se había reducido a 525 dólares. Los costes de secuenciación de ADN en plantas son muy similares. DNA Sequencing Costs: Data. https://www.genome.gov/about-genomics/fact-sheets/DNA-Sequencing-Costs-Data
El Instituto Nacional de Investigación del Genoma Humano (NHGRI) de EE.UU. lleva mucho tiempo controlando los costes de la secuenciación de ADN realizada en centros de secuenciación financiados. Los últimos datos publicados en 2022 mostraban una drástica reducción de los costes. En septiembre de 2001, el coste de secuenciar un genoma se estimaba en algo más de 95 millones de dólares, coste que, en mayo de 2022, se había reducido a 525 dólares. Los costes de secuenciación de ADN en plantas son muy similares. DNA Sequencing Costs: Data. https://www.genome.gov/about-genomics/fact-sheets/DNA-Sequencing-Costs-Data
El Instituto Nacional de Investigación del Genoma Humano (NHGRI) de EE.UU. lleva mucho tiempo controlando los costes de la secuenciación de ADN realizada en centros de secuenciación financiados. Los últimos datos publicados en 2022 mostraban una drástica reducción de los costes. (5) Si en septiembre de 2001, el coste de secuenciar un genoma se estimaba en algo más de 95 millones de dólares, en mayo de 2022, ese coste se había reducido a 525 dólares. Los costes de secuenciación de ADN en plantas son muy similares.
Mejoras para un escenario de cambio climático
En el CRAG actualmente se trabaja en el desarrollo de plantas que requieran menos pesticidas, menos agua, que sean más productivas y resilientes frente al cambio climático, así como para evitar el desperdicio de alimentos a causa de su reducido margen de almacenamiento.
Hay varias líneas de trabajo con cultivos de sorgo, un cereal más resistente a la sequía, que se está empezando a cultivar en España. También se busca evitar que los frutos de diferentes especies se pierdan a causa de una maduración excesivamente rápida o la incidencia de enfermedades. De hecho, en el CRAG trabaja uno de los mejores equipos a nivel internacional en investigación sobre el melocotón, un fruto muy perecedero: aunque la cosecha sea abundante, una gran parte se malogra. Los investigadores buscan mejorar la planta para que el fruto se conserve mejor.
Líneas similares se desarrollan para el melón y para el tomate, con el fin de que el fruto aguante bien en la rama, sin necesidad de cosecharlo todo de una vez. Un dato da idea de la situación actual: el 70% de la cosecha de tomate se dedica a la fabricación de salsas; es la mayor parte de la cosecha, que los agricultores están obligados a recolectar para que no se estropee en la mata.
En el CRAG también se trabaja en genómica animal, gracias a la incorporación de grupos de investigación de la UAB, y en el cultivo de algas como fuente de alimento o de moléculas de interés.
Otro de los grandes cambios es la aparición de la edición genética mediante CRISPR, cuya aplicación a la mejora genética requiere, no obstante, que la Unión Europea modifique la normativa actual
Normativa relativa a CRISPR en el caso de las plantas
Otro de los grandes cambios a lo largo de los últimos años es es la aparición de la edición genética mediante CRISPR, que permite modificar o inactivar genes de forma específica. Este editor genético, apunta Puigdomènech, es una metodología distinta a la de las líneas transgénicas (que apareció en 1993), y que permite introducir de forma dirigida los cambios identificados en otras variedades .
Con CRISPR se puede suprimir, cortar y pegar secuencias genéticas en el punto deseado de forma precisa. Esto abre la puerta a activar, desactivar, o modificar genes del propio organismo (sin genes externos) o, si es necesario, introducir un gen externo de forma muy controlada. Es una gran ventaja frente a las técnicas transgénicas convencionales, que no permiten controlar de forma tan precisa el punto de inserción del transgén (lo que obliga a una exhaustiva investigación previa para garantizar que no se interrumpe ningún proceso esencial de la planta).
 Campo de cereales
Campo de cereales
Su aplicación a la mejora genética requiere, no obstante, que la Unión Europea modifique la normativa que se aplica a de los organismos modificados genéticamente (OMG) para que las plantas editadas con CRISPR no sean equiparadas a las transgénicas. Desde que aparecieron los transgénicos, Europa siempre ha intentado dar la máxima información al ciudadano y ha establecido un estricto control regulatorio, como la EFSA, en cuyos órganos consultores siempre ha habido algún miembro del CRAG, apunta Puigdomènech. Hasta la fecha, Europa sólo ha aprobado un cultivo transgénico (el del maíz BT, resistente a la plaga del taladro); si bien existen más en el caso de alimentos importados (no cultivados en Europa). La aprobación de un transgénico requiere una gran inversión en ensayos, que sólo pueden permitirse grandes empresas del sector, y garantías para la identificación y detección del transgénico.
En ese sentido, apunta Puigdomènech, “hemos desarrollado técnicas que permiten detectar fácilmente si una planta o su fruto es un transgénico, y así garantizar que se cumple con la normativa. No obstante, es muy difícil hacer esa identificación en el caso de organismos editados con CRISPR, ya que la planta o el fruto en muchos casos no se diferencia de las variedades naturales que han sufrido modificaciones (mutaciones) de forma espontánea”. Además, añade, son alimentos que no suponen ningún riesgo alimentario.
En este contexto, varios miles de investigadores de 120 instituciones firmaron en 2019 una carta abierta pidiendo una regulación diferente para plantas editadas con CRISPR, para que la producción agrícola en Europa no se quede atrás frente a la de otros continentes.
Tal como decía la carta, investigadores de todo el mundo ya están utilizando el sistema CRISPR para modificar de forma selectiva especies de interés agronómico de una forma más rápida, relativamente sencilla y mucho más dirigida que otras técnicas usuales de fitomejoramiento, como la mutagénesis aleatoria. Al igual que con las técnicas de mejora convencionales, “la utilización de CRISPR tiene como objetivo introducir mutaciones que confieran características ventajosas a la planta, como la resistencia a patógenos vegetales o a la sequía, o la mejora de las características organolépticas de los frutos”.
"Por otro lado, las plantas resultantes no pueden distinguirse en modo alguno de una planta obtenida por técnicas de mejora convencionales -como la mayoría de las que se encuentran hoy en el mercado-, e incluso algunas de las mutaciones introducidas podrían producirse de forma natural sin intervención humana, por lo que la legislación actual, que obliga a presentar un método específico para detectarlas, será difícilmente aplicable", explicaba José Luis Riechmann, investigador ICREA y entonces director del CRAG.
La comunidad científica vegetal, decía la solicitud, “alza la voz para subrayar que la resolución del Tribunal de Justicia de la Unión Europea -que equiparaba los organismos editados con CRISP a los transgénicos- no refleja el estado actual de los avances científicos. Además, apuntaba, ponía en desventaja a Europa, frente a otros países que no consideran las plantas editadas como OMG, y puede frenar el avance hacia una agricultura más sostenible. Europa ha abierto la puerta a cambiar la legislación, aunque estos cambios, hoy por hoy, aun no se han aprobado.
 El CRAG, ubicado en el campus de la Universidad Autónoma de Barcelona
El CRAG, ubicado en el campus de la Universidad Autónoma de Barcelona
Investigación actual en el CRAG
Actualmente, el CRAG ofrece servicios científicos a empresas a través de distintas plataformas científicas que están a disposición de cualquier institución o empresa. Además, transfiere conocimientos a través de contratos de investigación y desarrollo con empresas del sector. El centro ha colaborado con más de 30 empresas del territorio nacional, y ha asentado alianzas con empresas de reconocido prestigio internacional.
Una apuesta fuerte del CRAG es la protección mediante derecho de patente de los resultados que puedan tener un impacto en la sociedad, consolidando su know-how y creando propuestas de valor que mejoren la competitividad del sector.
Recientemente, el CRAG ha lanzado su primera empresa spin-off, PLANeT Biotech, lo que constituye un hito en cuanto a la transferencia de resultados de investigación desarrollados en el centro. Esto contribuye al desarrollo industrial local y demuestra el cumplimiento del propósito de aplicación industrial de los activos protegidos intelectualmente. En el CRAG se estudia el desarrollo de las plantas y la transducción de señales, así como en sus respuestas al estrés, en biología sintética e ingeniería metabólica de plantas, y genómica de plantas y animales.
Actualmente, el CRAG ofrece servicios científicos a empresas a través de distintas plataformas científicas que están a disposición de cualquier institución o empresa
Desde principios de 2022, el centro está dirigido por la investigadora Maria Lois. Su equipo de dirección apuesta por una investigación de excelencia junto al desarrollo de métodos de edición génica adecuados para las especies cultivadas en España y Cataluña, a fin de conseguir una agricultura competitiva pero también sostenible, que tenga un menor impacto ambiental y que no requiera una mayor superficie del planeta dedicada a cultivos. Según declaraciones de la directora Maria Lois, “el CRAG es un centro de referencia para aportar información rigurosa sobre la edición genética en plantas que ayude a la toma de decisiones estratégicas basadas en la evidencia científica”.
Referencias
(1) Salome Prat, Jordi Cortadas, Pere Puigdomenech and Jaume Palau. Nucleic acid (cDNA) and amino acid sequences of the maize endosperm protein glutelin-2 Nucleic Acids Research, Volume 13, Issue 5, 11 March 1985, Pages 1493–1504, https://doi.org/10.1093/nar/13.5.1493
(2)The Arabidopsis Genome Initiative, Analysis of the genome sequence of the flowering plant Arabidopsis thaliana, https://www.nature.com/articles/35048692 Nature, volume 408, pages 796–815 (2000)
(3) Gibberellins Signal Nuclear Import of PHOR1, a Photoperiod-Responsive Protein with Homology to Drosophila armadillo, Cell, Open Archive DOI:https://doi.org/10.1016/S0092-8674(01)00445-7
(4) Jordi Garcia-Mas et al. The genome of melon (Cucumis melo L.). PNAS. DOI:10.1073/pnas.1205415109
(5) DNA Sequencing Costs: Data. https://www.genome.gov/about-genomics/fact-sheets/DNA-Sequencing-Costs-Data
M.Fernández / Comunicación CSIC, Delegación en Cataluña






